- La revista
- Air MUS12 Jordan 3LAB5 Gamma Blue Metallic Silver - MUS12 Jordan Air 200e Men Dc9836-200 - 200 - MUS12 Jordan Air 200e Men Dc9836
- Luxury Online Shop
- AspennigeriaShops , 5 Best Adidas Classic Trainers in 2024 , jam tangan adidas sport original
- Chaussures, sacs et vêtements
- Super Max Perfect Air Jordan 5 Women
- air jordan 4 white tech grey black fire red ct8527 100 release date
- adidas y 3 qasa high triple black
- air jordan 1 high og university blue 555088 134
- Gnarhunters Nike SB Dunk Low DH7756 010 Release Date On Foot
- nike dunk low university blue
- Presentación
- Políticas y proceso editorial
- Rigor científico y metodológico
- Producción y Administración
- Comités
- Normas presentación
- Consideraciones eticas
- Enviar artículo
- Hemeroteca
- Indexación
- Buscar
- Contacto
Farm Comunitarios. 2024 Apr 11;16(2):46-53. doi: 10.33620/FC.2173-9218.(2024).14
Diagnóstico por amplificación isotérmica de ácidos nucleicos. Oportunidad para la farmacia comunitaria
Introducción
En los últimos años hemos asistido a un cambio de paradigma en el diagnóstico de las enfermedades infecciosas. La tecnología ha evolucionado desde sistemas convencionales, relativamente complejos (ELISA, Western Blot, PCR) realizados en laboratorios especializados y con un tiempo de respuesta de horas a sistemas sencillos de autodiagnóstico (denominados POC, acrónimo del inglés Point of Care). Estos no requieren un equipamiento complejo, pueden ser realizados en la farmacia, consulta médica e incluso por el propio paciente y tienen un tiempo de respuesta de minutos (1). Según la Organización Mundial de la Salud (OMS), los test POC deben cumplir varias características sintetizadas en el acrónimo REASSURED que hace referencia a conectividad en tiempo real, facilidad de recolección de muestras, asequibilidad, sensibilidad, especificidad, facilidad de uso, rapidez y robustez (2).
La OMS considera a los test basados en la amplificación del material genético —como la PCR— como los de referencia en el diagnóstico de las enfermedades infecciosas (2). Sin embargo, estos sistemas se han resistido —hasta ahora— a su adaptación al paradigma de métodos tipo POC. Los cuatro tipos de test de diagnóstico de enfermedades infecciosas han alcanzado la categoría POC o están próximos a ella. Primeramente, los basados en la detección de proteínas propias del patógeno, denominados de antígenos que incluyen los ampliamente conocidos test rápidos de flujo lateral. La detección de anticuerpos frente al agente infeccioso —serológicos— serían el segundo tipo, usados para confirmar una infección pasada o en curso. Podríamos citar en tercer lugar la cuantificación de metabolitos inducidos por la infección como pueden ser la presencia de nitritos en infecciones de orina. Finalmente, disponemos de los que permiten detectar el material genético —ADN o ARN— comúnmente designadas como técnicas moleculares. La PCR estaría en esta categoría, pero no está considerada como un sistema POC por la necesidad de realizarse en un laboratorio y con equipamiento especializado (3).
La información práctica que proporcionan la detección de antígenos o ADN/ARN es similar, siendo un marcador específico de una infección activa (4,5). Los anticuerpos pueden complementar la información anterior, revelando una infección previa (IgG) o en curso (IgM) (5). Los test de metabolitos son muy útiles pero inespecíficos y no permiten discriminar un patógeno concreto (6,7). Los sistemas de diagnóstico molecular son significativamente más sensibles que los diagnósticos antigénicos, presentando un límite de detección de muy pocas copias del material genético diana. Además de la información cualitativa, los test moleculares también pueden ser cuantitativos, permitiendo monitorizar la evolución de la carga viral o bacteriana en el tiempo y en consecuencia la respuesta al tratamiento (8). Sin embargo, presentan varias desventajas, como son la necesidad de una purificación del material genético antes de la PCR, uso de un equipo más costoso, personal entrenado para realizar las pruebas y un lapso de tiempo más dilatado entre la toma de la muestra y el resultado final (9).
Esta revisión se centra en la descripción de los nuevos sistemas de diagnóstico molecular tipo POC disponibles en el mercado y que pueden ser fácilmente implementados en la farmacia comunitaria, con el potencial de ampliar la cartera de servicios farmacéuticos y contribuir de forma significativa a la mejora de la salud pública (10).
El conocimiento de las de nuevas técnicas de diagnóstico molecular diferentes de la PCR es relativamente desconocido. Sin embargo, las opciones disponibles son muy variadas y han alcanzado una madurez tecnológica suficiente para su uso masivo (11,12). La pandemia de SARS-CoV-2 ha contribuido a poner en el mercado test de diagnóstico que en algunos casos tenían varias décadas de uso exclusivamente a nivel de investigación.
Mercado de test isotérmicos ‘Point of Care’
Los datos disponibles de Comisión Europea sobre los test de diagnóstico molecular de SARS-CoV-2 son un indicativo de la implantación de las tecnologías isotérmicas comparadas con los sistemas convencionales basados en PCR para la detección de enfermedades infecciosas (13). Los test isotérmicos constituyen un porcentaje pequeño con respecto a los sistemas basados en PCR incluyendo los convencionales y los automatizados —cerca del nivel POC—. De los 304 test comercializados en Europa con marca CE (https://covid-19-diagnostics.jrc.ec.europa.eu/), sólo 25 son isotérmicos y de ellos 16 se corresponden con la tecnología LAMP (Loop-mediated isothermal amplification) (14).
El resto de tecnologías isotérmicas sólo tienen disponibles únicamente uno o dos test comerciales para la detección ultrasensible del SARS-CoV-2 estas incluyen la Cross-Priming Amplification (CPA), Helicase Dependent Amplification (HDA) (15), Smartamp (16), Strand Displacement Amplification (SDA) (17), y Nicking and Extension Amplification Reaction (NEAR) (18) (figura 1). Para otros patógenos también hay disponibles test de amplificación isotérmica, por ejemplo, tuberculosis (19), gripe (20), VIH (21), candida (22), enfermedades de transmisión sexual (gonorrea, clamidia, ureaplasma, micoplasma y papilomas) (23,24), entre otras.
En otro contexto, también relacionado con aplicaciones en farmacia comunitaria, recientemente se ha aprobado el uso de un test rápido de amplificación de ADN isotérmico para la detección de mutaciones vinculadas a toxicidad por aminoglucósidos (25).
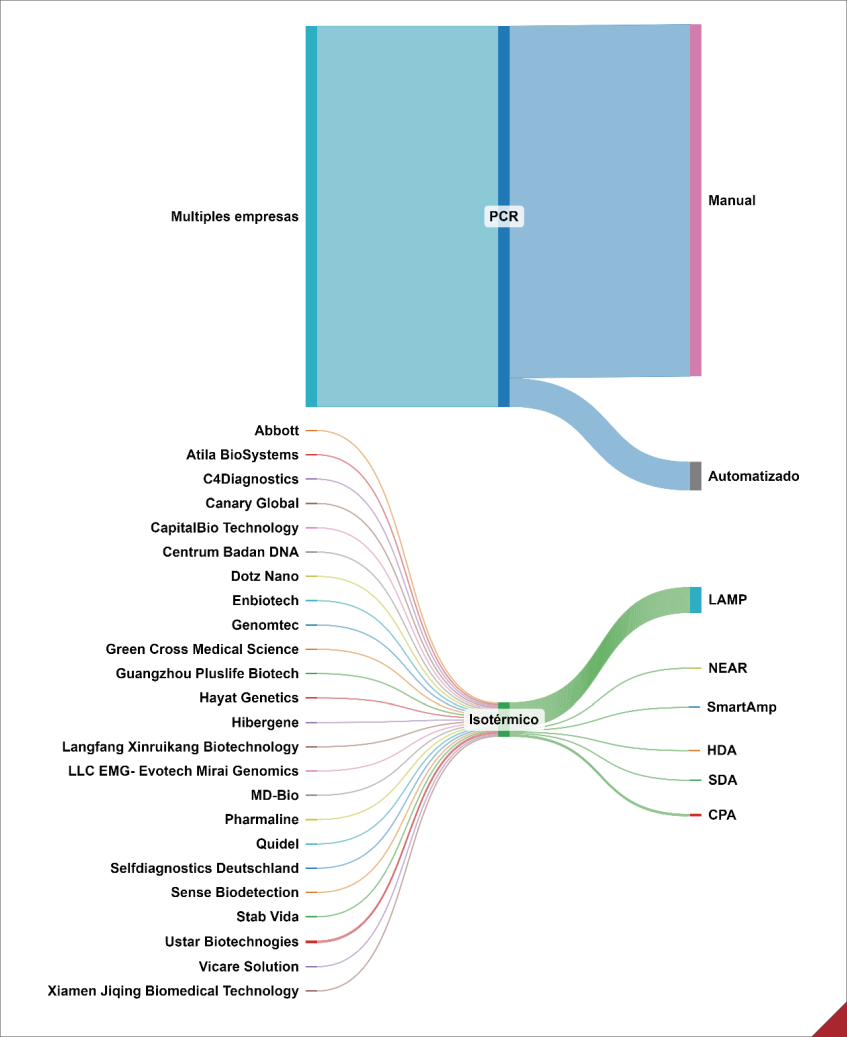
Figura 1 Sistemas de diagnóstico molecular por amplificación de ácidos nucleicos comercializados en Europa, con marca CE para diagnóstico del SARS-CoV-2. PCR manual (n=257), PCR automatizada próxima a nivel Point of Care (n=21). Isotérmicos (n=25) de los cuales LAMP (n=16), CPA (n=2) y HDA, SDA, NEAR y Smartamp (1 test disponible de cada tipo). Creado a partir de los datos de la Comisión Europea (13)
Fuente: elaboración propia
Características comunes de los test isotérmicos
La simplicidad del equipamiento y de la metodología necesaria para realizar los test isotérmicos, así como, la rapidez en su realización los hace adecuados para test tipo POC, especialmente en entornos con recursos limitados donde puede faltar equipo de laboratorio sofisticado (11). Sus particularidades podrían resumirse en los siguientes puntos:
- Reacción isotérmica. A diferencia de la PCR, que implica de 30 a 40 ciclos de tres temperaturas (desnaturalización del ADN, unión de cebadores y polimerización), los sistemas de amplificación isotérmica operan a una temperatura constante. Esto simplifica la instrumentación necesaria para la reacción ya que un simple termobloque que funciona a modo de baño maría es suficiente.
- Eficiencia de amplificación. Los sistemas isotérmicos pueden lograr una amplificación rápida y eficiente de los ácidos nucleicos. La amplificación es exponencial, lo que permite la detección de pequeñas cantidades de ácidos nucleicos del patógeno diana. La incorporación de una etapa de retrotranscripción anterior a la amplificación permite detectar virus de ARN.
- Diseño de cebadores. Al igual que otros métodos de amplificación de ácidos nucleicos, la amplificación isotérmica depende del uso de cebadores que se unen por complementariedad de bases a secuencias específicas del material genético del patógeno. El diseño de los cebadores es fundamental para la especificidad y eficiencia de la reacción. Esta es una de las principales limitaciones para estas tecnologías ya que en algunos casos requieren unos cebadores modificados que incorporan características específicas como regiones de auto apareamiento para formar bucles o bien sitios de reconocimiento de corte con nucleasas. Por ejemplo, las tecnologías LAMP y Smartamp generan estructuras en bucle como intermediarios en el proceso de amplificación. Estas estructuras en bucle contribuyen al alto rendimiento y velocidad de la reacción.
- Estos sistemas amplifican el ADN utilizando ADN polimerasas especializadas. Estas tienen capacidad de desplazamiento de la doble hebra del ADN, mediate la rotura de puentes de hidrógeno, desenrollándolo a medida que realizan la polimerización. Por ejemplo, la polimerasa Bst (aislada de la bacteria Bacillus stearothermophilus) se usa comúnmente en reacciones de amplificación isotérmica.
- Los métodos de detección del ADN amplificado varían, pero a menudo implican la medida en tiempo real de la reacción mediante fluorescencia, turbidez o colorimetría. Aunque el uso de un lector es necesario en la mayoría de los casos, en algunos sistemas comerciales, el diagnóstico se puede lograr mediante simple inspección visual.
Características específicas
LAMP- Loop-mediated isothermal amplification y Smartamp
Su característica fundamental es el uso de 4 cebadores, dos externos convencionales y dos internos que tienen complementariedad de bases con una región en el extremo 3´ de la cadena de ADN. Después de la unión de los cebadores, la ADN polimerasa incorpora nuevos nucleótidos y genera un intermediario de ADN monocatenario que se pliega sobre sí mismo mediante la formación de puentes de hidrógeno intracatenarios. Este intermediario es un nuevo sustrato para la ADN polimerasa que lo extiende nuevamente por incorporación de nucleótidos, generando un nuevo fragmento de ADN bicatenario. Los cebadores externos contribuyen a formar nuevo ADN diana para los cebadores internos. Este procedimiento genera fragmentos de ADN en tándem de varias kilobases de longitud en un proceso altamente eficiente, incluso dos órdenes de magnitud superior a un PCR convencional, una representación gráfica del proceso puede consultarse en la referencia Tomita N. et al (26). Las estimaciones indican que a partir de una sola copia de ADN/ARN pueden generarse hasta 109 copias en unos 30 minutos. La reacción tiene lugar a 65 ºC que es la temperatura óptima para la ADN polimerasa Bst. En el caso de detección de virus de ARN, la amplificación viene precedida de un paso de retrotranscripción de ARN a ADN complementario.
Una de las ventajas del sistema LAMP es que es muy resistente a inhibidores de la ADN polimerasa presentes en los fluidos biológicos, permite amplificar el material genético de un patógeno, sin necesidad purificación y a partir de todo tipo de muestras como saliva, esputo, orina, heces o incluso aguas residuales (27–30). Ha sido descrita una sensibilidad de 1 copia por µL y una especificidad mayor al 95 % en todos los estudios (11,12,27,31). Uno de los inconvenientes de LAMP es que no podía realizar amplificaciones de varias dianas simultáneamente, sin embargo, una modificación de la técnica denominada Omega de Atila Biosystems, sí lo permite (32). La mayoría de los test disponibles utilizan una medida de la fluorescencia a tiempo final para la determinación de la positividad para ello incorporan un agente intercalante que sólo es fluorescente cuando se incorpora a la doble hélice del ADN. También se puede utilizar la turbidimetría o colorimetría (tabla 1).
Tabla 1 Selección de test de diagnóstico basados en sistemas isotérmicos comercializados en Europa con marcado CE (33)
Una variante del método LAMP desarrollada en Japón y comercializada como Smartamp, también utiliza un conjunto de 5 cebadores que forman bucles de autoapareamiento (34,35). Un esquema del método puede consultarse en la publicación de Ishikawa y Hayashizaki (36). Actualmente, la empresa Evotech-Mirai Genomics utiliza la tecnología SmartAmp para COVID-19. En el diagnóstico de COVID-19, se ha comunicado una sensibilidad del 84 % y una especificidad del 95 % comparada con una PCR estándar (16) (tabla 1). Esta metodología también se ha empleado para el diagnóstico de tuberculosis (37), gripe (38) y en farmacogenética (36,39).
SDA-Strand Displacement Amplification y NEAR-Nicking and Extension Amplification Reaction
Originalmente desarrollada por la empresa Beckton-Dickinson (40), la técnica SDA requiere la combinación de 4 cebadores, dos externos convencionales y dos internos que contienen la secuencia de reconocimiento de corte de una nucleasa de cadena sencilla (Nt.BstNBI), también es necesaria una ADN polimerasa con desplazamiento de hebra. En una primera fase, con los cebadores externos se generan fragmentos de ADN monocatenario sobre los que hibridarán los cebadores internos. La polimerasa extenderá estos cebadores generando un ADN bicatenario que contiene sitios de corte por la nucleasa en ambos extremos. El corte de una de las hebras de ADN por la nucleasa genera un extremo 3´ libre en el ADN, que es sustrato de la polimerasa la cual produce un nuevo ADN bicatenario que hibridará con los cebadores reversos. Este proceso ocurre de forma continua, generando una gran cantidad de ADN fácilmente detectable mediante fluorescencia con agentes intercalantes. Un esquema del funcionamiento de esta técnica puede consultarse en la publicación de Leonardo S et al. (41).
Para COVID-19 el test tipo SDA tiene una sensibilidad del 97 % comparada con una PCR de referencia y una especificidad del 100 % (42,43). El límite de detección es muy bajo situándose entre 1-10 copias/µl (44). Además de su uso para COVID-19, SDA está aprobada para la detección de enfermedades de transmisión sexual (C. trachomatis, N. gonorrhoeae y HHV-1/2) (12,45).
HDA- Helicase Dependent Amplification
Esta tecnología es realmente sencilla, primeramente, el ADN se desnaturaliza por calor. Esto permite la unión de un par de cebadores a una secuencia diana complementaria. A continuación, una enzima denominada helicasa —en presencia de una proteína estabilizadora de ADN monocatenario— abre la doble hélice del ADN y una ADN polimerasa replica las dos cadenas. Reiteradamente, la ADN helicasa utiliza el ADN recién sintetizado como nuevos sustratos, iniciando una nueva ronda de replicación. Este hecho genera una reacción en cadena, que da como resultado una amplificación exponencial isotérmica de la secuencia de ácido nucleico específica. Un esquema del proceso puede encontrarse en el artículo de Vincent et al. (15). La tecnología de HDA ha sido comercializada en Europa por la empresa Quidel para el diagnóstico del SARS-CoV-2 (tabla 1). Tiene una sensibilidad del 95 % y especificidad del 99% con respecto a una PCR, situándose el límite de detección en 7,5 copias del virus (46). También se ha utilizado para la detección de VIH-1 (47), estreptococos del grupo A (48), Neisseria gonorrhoeae (49), Clostridium difficile (50) o Staphylococcus aureus (51).
CPA-Cross Priming Amplification
Al igual que las tecnologías LAMP y Smartamp, utiliza un conjunto de hasta 6 cebadores que forman bucles de autoapareamiento en un complejo intermediario que favorece la replicación exponencial del fragmento de ADN diana. Los detalles técnicos pueden consultarse en el artículo de Xu G et al. (52). Actualmente este sistema está comercializado por la empresa Ustar Biotechnologies en Europa para el diagnóstico del SARS-CoV-2 (tabla 1). Presenta una sensibilidad del 95 % comparada con la PCR y una especificidad del 98%, el límite de detección está en torno a 20 copias/µl. CPA también se ha incluido en test moleculares rápidos para la detección de otros patógenos como VIH, tuberculosis, gripe y enfermedades de transmisión sexual (53,54).
Conclusiones
Como hemos podido comprobar, existen una gran variedad de formatos y tecnologías que pueden adaptarse a la disponibilidad de casi cualquier entorno desde laboratorios de investigación o clínicos hasta hogares, pasando por las farmacias.
Las tecnologías isotérmicas de autodiagnóstico o POC constituyen una alternativa competitiva a la PCR convencional realizada en laboratorios especializados o centros de referencia. El coste-tiempo desde la toma de muestra a los resultados, así como la facilidad para su implementación en farmacias comunitarias, constituye un potencial nicho de mercado para las mismas. A pesar de todo ello, el volumen de las pruebas isotérmicas que se realizan es relativamente reducido y muy desconocido en nuestro entorno.
Las farmacias constituyen uno de los puntales de la atención sanitaria primaria, especialmente en momentos de saturación de urgencias y ambulatorios. Por ello, contar con un arsenal diagnóstico de fácil implementación y uso constituye una herramienta esencial para el control y manejo de las enfermedades infecciosas, tal como hemos podido comprobar durante la pandemia del COVID-19.
Las tecnologías isotérmicas están en evolución permanente y en los próximos años asistiremos —con toda probabilidad— a un crecimiento exponencial del uso de las mismas y a nuevos desarrollos tecnológicos que simplifiquen y abaraten aún más el coste por determinación. El conocimiento de las mismas es una estrategia esencial de vigilancia tecnológica e inteligencia competitiva de las oficinas de farmacia.
REFERENCIAS BIBLIOGRÁFICAS
1. Klepser M, Koski RR. Molecular point-of-care testing in the community pharmacy setting: current status and future prospects [Internet]. Vol. 22, Expert Review of Molecular Diagnostics. Taylor and Francis Ltd.; 2022 [cited 2023 Jan 24]. p. 923–36. Disponible en: https://pubmed.ncbi.nlm.nih.gov/36269237/
2. Land KJ, Boeras DI, Chen XS, Ramsay AR, Peeling RW. REASSURED diagnostics to inform disease control strategies, strengthen health systems and improve patient outcomes. Nat Microbiol [Internet]. 2019 Jan 1 [cited 2023 Feb 21];4(1):46–54. Disponible en: https://pubmed.ncbi.nlm.nih.gov/30546093/
3. Weber NC, Klepser ME, Akers JM, Klepser DG, Adams AJ. Use of CLIA-waived point-of-care tests for infectious diseases in community pharmacies in the United States. Expert Rev Mol Diagn [Internet]. 2016 Feb 1 [cited 2023 Jan 24];16(2):253–64. Disponible en: https://pubmed.ncbi.nlm.nih.gov/26560318/
4. Dinnes J, Deeks JJ, Berhane S, Taylor M, Adriano A, Davenport C, et al. Rapid, point-of-care antigen and molecular-based tests for diagnosis of SARS-CoV-2 infection. Cochrane database Syst Rev [Internet]. 2021 Mar 24 [cited 2024 Mar 14];3(3). Disponible en: https://pubmed.ncbi.nlm.nih.gov/33760236/
5. Corman VM, Haage VC, Bleicker T, Schmidt ML, Mühlemann B, Zuchowski M, et al. Comparison of seven commercial SARS-CoV-2 rapid point-of-care antigen tests: a single-centre laboratory evaluation study. The Lancet Microbe [Internet]. 2021 Jul 1 [cited 2023 Feb 6];2(7):e311–9. Disponible en: https://pubmed.ncbi.nlm.nih.gov/33846704/
6. Tomlinson E, Jones HE, James R, Cooper C, Stokes C, Begum S, et al. Clinical effectiveness of point of care tests for diagnosing urinary tract infection: a systematic review. Clin Microbiol Infect [Internet]. 2024 Feb 1 [cited 2024 Mar 14];30(2):197–205. Disponible en: https://pubmed.ncbi.nlm.nih.gov/37839580/
7. Scagnolari C, Turriziani O, Monteleone K, Pierangeli A, Antonelli G. Consolidation of molecular testing in clinical virology [Internet]. Vol. 15, Expert Review of Anti-Infective Therapy. Taylor and Francis Ltd; 2017 [cited 2024 Mar 14]. p. 387–400. Disponible en: https://pubmed.ncbi.nlm.nih.gov/28002969/
8. Artika IM, Dewi YP, Nainggolan IM, Siregar JE, Antonjaya U. Real-Time Polymerase Chain Reaction: Current Techniques, Applications, and Role in COVID-19 Diagnosis. Genes (Basel) [Internet]. 2022 Dec 1 [cited 2024 Mar 14];13(12). Disponible en: https://pubmed.ncbi.nlm.nih.gov/36553654/
9. Phan T, Nagaro K. Diagnostic Tests for COVID-19. Adv Exp Med Biol [Internet]. 2021 [cited 2024 Mar 14];1318:403–12. Disponible en: https://pubmed.ncbi.nlm.nih.gov/33973191/
10. Maidment I, Young E, MacPhee M, Booth A, Zaman H, Breen J, et al. Rapid realist review of the role of community pharmacy in the public health response to COVID-19. BMJ Open [Internet]. 2021 Jun 16 [cited 2023 Jan 24];11(6). Disponible en: https://pubmed.ncbi.nlm.nih.gov/34135054/
11. García-Bernalt Diego J, Fernández-Soto P, Muro A. The Future of Point-of-Care Nucleic Acid Amplification Diagnostics after COVID-19: Time to Walk the Walk. Int J Mol Sci [Internet]. 2022 Nov 1 [cited 2024 Jan 7];23(22). Disponible en: https://pubmed.ncbi.nlm.nih.gov/36430586/
12. Oliveira BB, Veigas B, Baptista PV. Isothermal Amplification of Nucleic Acids: The Race for the Next “Gold Standard.” Front Sensors [Internet]. 2021 Sep 28 [cited 2023 Jan 19];2:14. Disponible en: https://www.frontiersin.org/articles/10.3389/fsens.2021.752600/full
13. In Vitro Diagnostics EUAs - Molecular Diagnostic Tests for SARS-CoV-2 | FDA [Internet]. [cited 2023 Jan 9]. Disponible en: https://www.fda.gov/medical-devices/coronavirus-disease-2019-covid-19-emergency-use-authorizations-medical-devices/in-vitro-diagnostics-euas-molecular-diagnostic-tests-sars-cov-2#individual-molecular
14. Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, et al. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 2000;28(12):e63. https://pubmed.ncbi.nlm.nih.gov/10871386/
15. Vincent M, Xu Y, Kong H. Helicase-dependent isothermal DNA amplification. EMBO Rep [Internet]. 2004 Aug [cited 2023 Jan 5];5(8):795–800. Disponible en: https://pubmed.ncbi.nlm.nih.gov/15247927/
16. Asai N, Nakamura A, Sakanashi D, Koita I, Ohashi W, Kawamoto Y, et al. Comparative study of SmartAmp assay and reverse transcription-polymerase chain reaction by saliva specimen for the diagnosing COVID-19. J Infect Chemother [Internet]. 2022 Jan 1 [cited 2023 Jan 27];28(1):120–3. Disponible en: https://pubmed.ncbi.nlm.nih.gov/34580006/
17. Toley BJ, Covelli I, Belousov Y, Ramachandran S, Kline E, Scarr N, et al. Isothermal strand displacement amplification (iSDA): a rapid and sensitive method of nucleic acid amplification for point-of-care diagnosis. Analyst [Internet]. 2015 Nov 21 [cited 2023 Feb 21];140(22):7540–9. Disponible en: https://pubmed.ncbi.nlm.nih.gov/26393240/
18. Van Ness J, Van Ness LK, Galas DJ. Isothermal reactions for the amplification of oligonucleotides. Proc Natl Acad Sci U S A [Internet]. 2003 Apr 15 [cited 2024 Jan 6];100(8):4504–9. Disponible en: https://pubmed.ncbi.nlm.nih.gov/12679520/
19. Deng S, Sun Y, Xia H, Liu Z, Gao L, Yang J, et al. Accuracy of Commercial Molecular Diagnostics for the Detection of Pulmonary Tuberculosis in China: A Systematic Review. Sci Rep [Internet]. 2019 Dec 1 [cited 2023 Jan 27];9(1). Disponible en: https://pubmed.ncbi.nlm.nih.gov/30872692/
20. Young S, Illescas P, Nicasio J, Sickler JJ. Diagnostic accuracy of the real-time PCR cobas® Liat® Influenza A/B assay and the Alere i Influenza A&B NEAR isothermal nucleic acid amplification assay for the detection of influenza using adult nasopharyngeal specimens. J Clin Virol [Internet]. 2017 Sep 1 [cited 2024 Jan 6];94:86–90. Disponible en: https://pubmed.ncbi.nlm.nih.gov/28772170/
21. Mourez T, Delaugerre C, Vray M, Lemée V, Simon F, Plantier JC. Comparison of the bioMérieux NucliSENS EasyQ HIV-1 v2.0-HIV-1 RNA quantification assay versus Abbott RealTime HIV-1 and Roche Cobas TaqMan HIV-1 v2.0 on current epidemic HIV-1 variants. J Clin Virol [Internet]. 2015 Oct 1 [cited 2024 Jan 6];71:76–81. Disponible en: https://pubmed.ncbi.nlm.nih.gov/26318912/
22. Hernández Felices FJ, Tormo Palop N, Salvador García C, Mulet Bayona JV, Guna Serrano MR, Gimeno Cardona C. Evaluation of Eazyplex® LAMP test for fast Candida auris direct detection of colonized patients. Mycoses [Internet]. 2023 Jan 1 [cited 2024 Jan 6]; Disponible en: https://pubmed.ncbi.nlm.nih.gov/37860835/
23. Tang YW, Lozano L, Chen X, Querec TD, Katabi N, Moreno-Docón A, et al. An Isothermal, Multiplex Amplification Assay for Detection and Genotyping of Human Papillomaviruses in Formalin-Fixed, Paraffin-Embedded Tissues. J Mol Diagn [Internet]. 2020 Mar 1 [cited 2023 Jan 10];22(3):419–28. Disponible en: https://pubmed.ncbi.nlm.nih.gov/31978559/
24. Esitgen Germaner ET, Wassill L, Dichtl K, Roider J, Seybold U. Rapid point of care testing for four bacterial sexually transmitted infections using the portable isothermal loop-mediated nucleic acid amplification eazyplex platform. Infection [Internet]. 2023 Aug 1 [cited 2024 Jan 6];51(4):1141–5. Disponible en: https://pubmed.ncbi.nlm.nih.gov/36641506/
25. McDermott JH, Mahaveer A, James RA, Booth N, Turner M, Harvey KE, et al. Rapid Point-of-Care Genotyping to Avoid Aminoglycoside-Induced Ototoxicity in Neonatal Intensive Care. JAMA Pediatr [Internet]. 2022 May 1 [cited 2024 Jan 6];176(5):486. Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8938898/
26. Tomita N, Mori Y, Kanda H, Notomi T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nat Protoc [Internet]. 2008 Apr [cited 2024 Jan 7];3(5):877–82. Disponible en: https://pubmed.ncbi.nlm.nih.gov/18451795/
27. Kidd SP, Burns D, Armson B, Beggs AD, Howson ELA, Williams A, et al. Reverse-Transcription Loop-Mediated Isothermal Amplification Has High Accuracy for Detecting Severe Acute Respiratory Syndrome Coronavirus 2 in Saliva and Nasopharyngeal/Oropharyngeal Swabs from Asymptomatic and Symptomatic Individuals. J Mol Diagn [Internet]. 2022 Apr 1 [cited 2024 Jan 7];24(4):320–36. Disponible en: https://pubmed.ncbi.nlm.nih.gov/35121140/
28. Ongerth JE, Danielson RE. RT qLAMP-Direct Detection of SARS-CoV-2 in Raw Sewage. J Biomol Tech [Internet]. 2021 Sep 1 [cited 2024 Jan 7];32(3):206–13. Disponible en: https://pubmed.ncbi.nlm.nih.gov/35027878/
29. Beggs AD, Manzoor SE, Zaman S, Whalley C, Inglis D, Bosworth A, et al. Multi-modality detection of SARS-CoV-2 in faecal donor samples for transplantation and in asymptomatic emergency surgical admissions. F1000Research [Internet]. 2021 [cited 2024 Jan 7];10. Disponible en: https://pubmed.ncbi.nlm.nih.gov/34367617/
30. Rödel J, Egerer R, Suleyman A, Sommer-Schmid B, Baier M, Henke A, et al. Use of the variplexTM SARS-CoV-2 RT-LAMP as a rapid molecular assay to complement RT-PCR for COVID-19 diagnosis. J Clin Virol [Internet]. 2020 Nov 1 [cited 2024 Jan 7];132. Disponible en: https://pubmed.ncbi.nlm.nih.gov/32891938/
31. Baba MM, Bitew M, Fokam J, Lelo EA, Ahidjo A, Asmamaw K, et al. Diagnostic performance of a colorimetric RT -LAMP for the identification of SARS-CoV-2: A multicenter prospective clinical evaluation in sub-Saharan Africa. EClinicalMedicine [Internet]. 2021 Oct 1 [cited 2024 Jan 7];40. Disponible en: https://pubmed.ncbi.nlm.nih.gov/34476394/
32. WO2017205510A1 - Omega amplification - Google Patents [Internet]. [cited 2023 Jan 10]. Disponible en: https://patents.google.com/patent/WO2017205510A1/en
33. Joint Research Centre EC. COVID-19 In Vitro Diagnostic Devices and Test Methods Database [Internet]. 2022 [cited 2023 Jan 3]. Disponible en: https://covid-19-diagnostics.jrc.ec.europa.eu/
34. Mitani Y, Lezhava A, Kawai Y, Kikuchi T, Oguchi-Katayama A, Kogo Y, et al. Method of amplifying nucleic acid and method of detecting mutated nucleic acid using the same [Internet]. European Patent Office; EP1712618B1, 2004 [cited 2023 Jan 16]. Disponible en: https://patents.google.com/patent/EP1712618B1/ko
35. Mitani Y, Lezhava A, Kawai Y, Kikuchi T, Oguchi-Katayama A, Kogo Y, et al. Rapid SNP diagnostics using asymmetric isothermal amplification and a new mismatch-suppression technology. Nat Methods [Internet]. 2007 Mar 18 [cited 2023 Jan 4];4(3):257–62. Disponible en: https://www.nature.com/articles/nmeth1007
36. Ishikawa T, Hayashizaki Y. Clinical SNP detection by the SmartAmp method. Methods Mol Biol [Internet]. 2013 [cited 2024 Jan 8];1015:55–69. Disponible en: https://pubmed.ncbi.nlm.nih.gov/23824848/
37. Chen CA, Ho NYJ, Hsiao HY, Lin SS, Lai PL, Tsai TT. Smartphone-assisted fluorescence-based detection of sunrise-type smart amplification process and a 3D-printed ultraviolet light-emitting diode device for the diagnosis of tuberculosis. Biosens Bioelectron [Internet]. 2024 Jan 15 [cited 2024 Jan 8];244. Disponible en: https://pubmed.ncbi.nlm.nih.gov/37918047/
38. Kawai Y, Kimura Y, Lezhava A, Kanamori H, Usui K, Hanami T, et al. One-step detection of the 2009 pandemic influenza A(H1N1) virus by the RT-SmartAmp assay and its clinical validation. PLoS One [Internet]. 2012 Jan 25 [cited 2024 Jan 8];7(1). Disponible en: https://pubmed.ncbi.nlm.nih.gov/22295077/
39. Aw W, Lezhava A, Andoh A, Tanaka H, Hayashizaki Y, Ishikawa T. The SmartAmp method: rapid detection of SNPs in thiopurine S-methyltransferase and ABC transporters ABCC4 and ABCG2. Curr Drug Metab [Internet]. 2012 Aug 2 [cited 2024 Jan 9];13(7):968–77. Disponible en: https://pubmed.ncbi.nlm.nih.gov/22591349/
40. Walker GT. Strand displacement amplification. Patent [Internet]. US5712124A, 1995 [cited 2023 Jan 9]. p. US5712124A. Disponible en: https://patents.google.com/patent/US5712124A/en
41. Leonardo S, Toldrà A, Campàs M. Biosensors Based on Isothermal DNA Amplification for Bacterial Detection in Food Safety and Environmental Monitoring. Sensors (Basel) [Internet]. 2021 Jan 2 [cited 2024 Jan 8];21(2):1–24. Disponible en: https://pubmed.ncbi.nlm.nih.gov/33467078/
42. Lamble HJ, Lloyd D. Nucleic acid detection method [Internet]. United States; US20210040571A1, 2020 [cited 2023 Jan 4]. p. US20210040571A1. Disponible en: https://patents.google.com/patent/US20210040571A1/en
43. Veros COVID-19 | Sense [Internet]. [cited 2023 Jan 16]. Disponible en: https://sense-bio.com/veros-covid-19/
44. Hellyer TJ, Nadeau JG. Strand displacement amplification: a versatile tool for molecular diagnostics. Expert Rev Mol Diagn [Internet]. 2004 Mar [cited 2023 Jan 27];4(2):251–61. Disponible en: https://pubmed.ncbi.nlm.nih.gov/14995911/
45. Lee H, Lee H, Hwang SH, Jeong W, Kim DE. Detection of SARS-CoV-2 RNA through tandem isothermal gene amplification without reverse transcription. Anal Chim Acta [Internet]. 2022 Jun 15 [cited 2023 Jan 20];1212. Disponible en: https://pubmed.ncbi.nlm.nih.gov/35623783/
46. Quidel. Solana SARS-CoV-2 Assay [Internet]. 2020. Disponible en: https://www.fda.gov/media/144759/download
47. Tang W, Chow WHA, Li Y, Kong H, Tang YW, Lemieux B. Nucleic acid assay system for tier II laboratories and moderately complex clinics to detect HIV in low-resource settings. J Infect Dis [Internet]. 2010 [cited 2024 Jan 8];201 Suppl. Disponible en: https://pubmed.ncbi.nlm.nih.gov/20225946/
48. Pumford EA, Lu J, Spaczai I, Prasetyo ME, Zheng EM, Zhang H, et al. Developments in integrating nucleic acid isothermal amplification and detection systems for point-of-care diagnostics. Biosens Bioelectron [Internet]. 2020 Dec 15 [cited 2023 Jan 16];170. Disponible en: https://pubmed.ncbi.nlm.nih.gov/33035900/
49. Doseeva V, Forbes T, Wolff J, Khripin Y, O’Neil D, Rothmann T, et al. Multiplex isothermal helicase-dependent amplification assay for detection of Chlamydia trachomatis and Neisseria gonorrhoeae. Diagn Microbiol Infect Dis [Internet]. 2011 Dec [cited 2024 Jan 8];71(4):354–65. Disponible en: https://pubmed.ncbi.nlm.nih.gov/22000085/
50. Eckert C, Holscher E, Petit A, Lalande V, Barbut F. Molecular test based on isothermal helicase-dependent amplification for detection of the Clostridium difficile toxin a gene. J Clin Microbiol [Internet]. 2014 [cited 2024 Jan 8];52(7):2386–9. Disponible en: https://pubmed.ncbi.nlm.nih.gov/24759714/
51. Chen X, Wu X, Gan M, Xu F, He L, Yang D, et al. Rapid detection of Staphylococcus aureus in dairy and meat foods by combination of capture with silica-coated magnetic nanoparticles and thermophilic helicase-dependent isothermal amplification. J Dairy Sci [Internet]. 2015 Mar 1 [cited 2024 Jan 8];98(3):1563–70. Disponible en: https://pubmed.ncbi.nlm.nih.gov/25547304/
52. Xu G, Hu L, Zhong H, Wang H, Yusa SI, Weiss TC, et al. Cross priming amplification: mechanism and optimization for isothermal DNA amplification. Sci Rep [Internet]. 2012 [cited 2023 Jan 16];2. Disponible en: https://pubmed.ncbi.nlm.nih.gov/22355758/
53. Chen G, Yu J, Chen H, Cen K, Zhou Y, You Q, et al. EasyNAT MP Assay: A Simple, Rapid, and Low-Cost Method to Detect Mycoplasma pneumoniae Using Cross-Priming Amplification Technology. Mol Diagn Ther [Internet]. 2022 May 1 [cited 2024 Jan 8];26(3):345–52. Disponible en: https://pubmed.ncbi.nlm.nih.gov/35430704/
54. You Q. Cross priming amplification of target nucleic acids. Patent [Internet]. EP2382330A1, 2010. p. EP2382330A1. Disponible en: https:// patents.google.com/patent/EP2382330A1/un
Editado por: © SEFAC. Sociedad Española de Farmacia Clínica, Familiar y Comunitaria.
© Copyright SEFAC. Sociedad Española de Farmacia Clínica, Familiar y Comunitaria. Este artículo está disponible en la url https://www.farmaceuticoscomunitarios.org/ Este trabajo está bajo la licencia Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International License. Las imágenes u otro material de terceros en este artículo se incluyen en la licencia Creative Commons del artículo, a menos que se indique lo contrario en la línea de crédito. Si el material no está incluido en la licencia Creative Commons, los usuarios deberán obtener el permiso del titular de la licencia para reproducir el material. Para ver una copia de esta licencia, visite https://creativecommons.org/licenses/by-nc-nd/4.0/deed.es_ES


